关于CGH探针密度的指导 |
|
最低的探针数目取决于畸变区的大小、做出畸变判断所需的探针数(例如:1,2,3或以上)和实验的整体噪音。DLRSD(derivative log ratio spread, probe-to-probe log ratio noise)质量控制标准是一个对于安捷伦aCGH实验的整体噪音的比较好的估计,这和DNA的质量密切相关。 至于最大探针数,在某一区域放置更多探针并不一定帮助确认小范围的畸变(请参见Ylstra 等人所著的(Nucleic Acids Research, 2006, Vol. 34, No. 2 for a discussion on functional resolution of CGH experiments)。首先,如果创建一个密度极高的设计,它必然包含一些分数较低的探针。安捷伦HD-CGH数据库中的所有探针都有一个预期的表现评分(基于解链温度,GC含量,一个发夹deltaG,序列复杂性,以及衡量与参照基因组的其余部分的相似度的标准)。eArray的成对还原算法(pair-wise reduction algorithm)将基于用户选择的平均高密度探针间距、或者高密度探针总数来选择最佳的探针。在一个密度极高的设计的情况下,成对还原算法将不再能够返回最好的探针,而将返回所有或大部份可用探针。请查看常见问题“什么是CGH探针评分?”来获取更多信息。
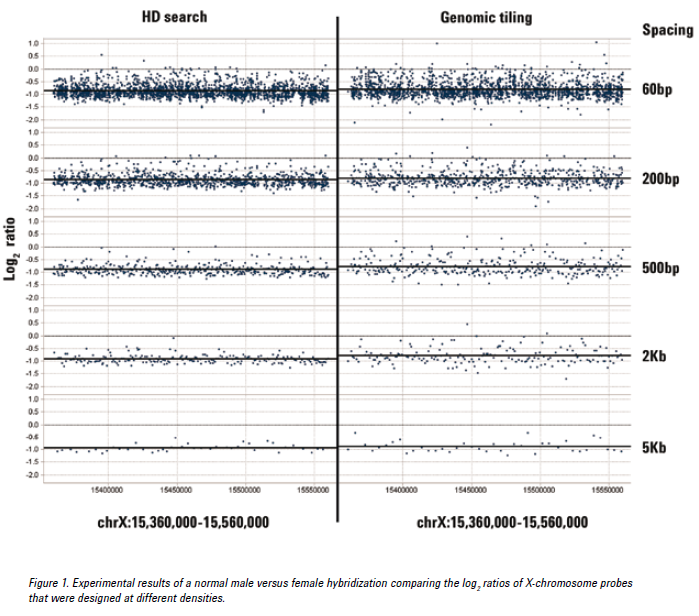
此外,密度极高的设计有可能使杂交产生不良的结果。安捷伦标准DNA标记流程,产生大约200nt的DNA判断。在一个密度极高的设计情况下,阵列上的探针将相互竞争相同的杂交片段。 在一小片区域放置过多探针将降低的信号的强度,可能产生有噪声的数据。安捷伦不建议使用低于150 - 200bp的探针间距。
图中的示例是使用高密度查找或基因组叠瓦式设计而获取的不同密度探针的CGH数据。间距为200bp的高密度查找获得的探针,产生的对数比非常接近于预期值-1。
